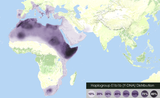
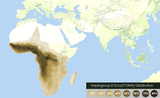
Haplogroupe E
| Date d'origine | il y a 73 000 ans ou 69 000 ans |
|---|---|
| Place d'origine | Afrique de l'Est ou Eurasie |
| Ancêtre | DE |
| Descendants | E-M75, E-P147 |
| Mutations définies | L339, L614, M40/SRY4064/SRY8299, M96, P29, P150, P152, P154, P155, P156, P162, P168, P169, P170, P171, P172, P173, P174, P175, P176 |
En génétique humaine, l’haplogroupe E (E-M96) est un haplogroupe du chromosome Y. Il s'agit de l'une des deux branches principales de l'haplogroupe plus ancien DE, l'autre étant l'haplogroupe D. Le clade E-M96 est divisé en deux sous-clades principaux: le plus commun E-P147 et le moins commun E-M75.
Distribution géographique
[modifier | modifier le code]Il est l'haplogroupe le plus commun en Afrique, où il serait apparu il y a au moins 69 000[1] ou 73 000 ans[2]. Cet haplogroupe se retrouve surtout en Afrique, au Proche-Orient mais également en Europe chez les Albanais entre 40 et 50 %, principalement dans le Sud et à l'Est à des fréquences plus faibles[3].
Origine
[modifier | modifier le code]L'origine géographique de cet haplogroupe est discutée, et il existe plusieurs hypothèses :
En 2001, Underhill avançait que l'haplogroupe E était probablement présent dans la région du Sahara, à une période où celui-ci n'était pas un désert (c'est-à-dire il y a environ 45 000 ans). Quand le Sahara a commencé à se désertifier, les habitants de la région en sont partis pour s'installer dans des zones plus favorables. C'est à cette époque que cet haplogroupe s'est divisé en plusieurs sous-groupes. Underhill proposait en 2001 l'est de l'Afrique comme lieu d'origine de cet haplogroupe[4].
En 2008, une étude de Karafet, dont Hammer était co-auteur, a fait valoir qu'une origine africaine était plus probable. Cette étude a considérablement réorganisé les estimations de temps conduisant à de nouvelles interprétations sur l'origine géographique des anciens sous-clades[5]. Entre autres choses, cet article proposait un âge beaucoup plus avancé pour l'haplogroupe E-M96 que ce qui avait été envisagé précédemment, lui donnant un âge similaire à l'haplogroupe D et à DE lui-même, ce qui signifie qu'il n'y a plus de raison forte de le voir comme une émanation de DE, qui a dû se produire longtemps après sa création et son entrée en Asie[5].
En 2015, Poznik et Underhill ont soutenu que l'haplogroupe E aurait surgi en dehors de l'Afrique. Ce modèle de ségrégation géographique au sein du clade CT ne nécessiterait qu'un seul échange d'haplogroupe continental (E vers l'Afrique), au lieu de trois (D, C et F) vers l'Asie. Le moment choisi pour ce retour présumé en Afrique, entre l'émergence de l'haplogroupe E et sa différenciation en Afrique de 58 000 ans, est conforme aux propositions fondées sur des données relatives au chromosome non-Y, de flux génétiques abondants entre l'Afrique et l'Arabie entre 50 et 80 000 ans[6].
En 2015, Trobetta et al. a suggéré une origine est-africaine pour l'haplogroupe E, déclarant : « notre analyse phylogéographique, basée sur des milliers d'échantillons dans le monde, suggère que le rayonnement de l'haplogroupe E a commencé à environ 58 ka, quelque part en Afrique subsaharienne, avec une probabilité postérieure plus élevée (0,73) pour une origine est-africaine »[7].
Un centre d'origine et de dispersion eurasien pour l'haplogroupe E a également été émis en 2018 sur la base de l'âge similaire de l'haplogroupe parent du clade, DE, et de l'haplogroupe L3 de l'ADNmt. Selon cette hypothèse, il a donc été proposé que, après une migration initiale extra-africaine d'Homo sapiens il y a autour de 125 000 ans, des hommes ayant un haplogroupe E eurasien soient revenus depuis le lieu d'origine de l'haplogroupe paternel en Eurasie autour de 70 000 ans, ainsi que des femmes porteuses de l'haplogroupe mitochondrial L3, dont on suppose également qu'il est originaire d'Eurasie. On suggère alors que ces nouvelles lignées eurasiennes ont largement remplacé les anciennes lignées africaines autochtones masculines et féminines[8].
Une étude de 2019 réalisée par Haber et al. soutient une origine africaine pour l'haplogroupe E (basé en partie sur une analyse de la structure phylogénétique du chromosome Y, des temps de divergence des haplogroupes et de l'haplogroupe D0 récemment découvert chez trois Nigérians, une branche supplémentaire de la lignée DE divergeant tôt de la branche D). Les auteurs soutiennent une origine africaine pour DE (haplogroupe parent de E), E et D0 étant également originaires d’Afrique, ainsi qu'une migration hors d'Afrique des trois lignées (C, D et FT) qui forment maintenant la grande majorité des chromosomes Y non-africains. Les dates de divergence précoces pour DE, E et D0, telles que trouvées dans l'étude, qui sont déterminées à précéder la migration extra-africaine d'Eurasiens ancestraux, sont également considérées par les auteurs comme supportant une origine africaine pour ces haplogroupes[2].
Sous-groupes
[modifier | modifier le code]-
Distribution de l'haplogroupe E1b1b.
-
Distribution de l'haplogroupe E1b1a.
E1a
[modifier | modifier le code]E1a (M33) présent au Mali.
E1b1
[modifier | modifier le code]E1b1, de loin le plus fréquent, est divisé en deux sous-groupes :
E1b1a
[modifier | modifier le code]L'haplogroupe E1b1a, caractérisé par le marqueur M2, est prévalent (70-100 %) chez les populations de l'Afrique de l'Ouest, l'Afrique Centrale, l'Afrique du Sud, et l'Afrique Sud-Est.
E1b1b (E-M35)
[modifier | modifier le code]
Diffusion de l'haplogroupe E1b1b.
L'haplogroupe E1b1b (ou E-M215, anciennement E3b) est caractérisé par le marqueur M215.
Son sous-groupe dominant est l'haplogroupe E1b1b1 (ou E-M35), caractérisé par le marqueur M35. Il est apparu pour la première fois au Levant il y a approximativement 22 000 ans, et s'est dispersé à travers le Proche-Orient et l'Afrique pendant le Néolithique. Il représente la plus récente migration majeure d'Afrique vers l'Europe.
E1b1b1 est prévalent chez les populations Afro-Asiatiques : Berbères, Coptes, Couchites et Sémites. Les populations européennes qui possèdent l'haplogroupe E1b1b1 sont pour la plupart originaires du pourtour méditerranéen. Elles auraient reçu l'influence génétique de l'Afrique du Nord et du Proche-Orient. Cet haplogroupe aurait été amené en Europe par des populations interagissant directement avec le Proche-Orient (V13). Ces dernières populations sont venues du Levant, via l'Asie Mineure. Ce sous-clade fait partie des haplogroupes des populations qui ont apporté l'agriculture en Europe au Néolithique.
Arbre phylogénétique
[modifier | modifier le code]Les noms de sous-groupes changeant fréquemment, l'arbre ci-dessous utilise les marqueurs principaux afin de distinguer les différents sous-groupes à l'intérieur du groupe principal E1b1b (ISOGG 2013).
- E1b1b (M215)
- E1b1b (M35.1)
- E1b1b (V68)
- E1b1b (M78)
- E1b1b (V12)
- E1b1b (V12 *)
- E1b1b (M224)
- E1b1b (V32)
- E1b1b (V13)
- E1b1b (L17)
- E1b1b (L143)
- E1b1b (V22)
- E1b1b (M148)
- E1b1b (V19)
- E1b1b (V65)
- E1b1b (V12)
- E1b1b (M78)
- E1b1b (Z827)
- E1b1b (V257)
- E1b1b (M81)
- E1b1b (M183)
- E1b1b (M81)
- E1b1b (Z830)
- E1b1b (M123)
- E1b1b (M34)
- E1b1b (M84)
- E1b1b (M34)
- E1b1b (M293)
- E1b1b (V42)
- E1b1b (M123)
- E1b1b (V257)
- E1b1b (V6)
- E1b1b (V92)
- E1b1b (V68)
- E1b1b (V16)
- E1b1b (M35.1)
E-M78
[modifier | modifier le code]E-M78 (anciennement E1b1b1a) est apparu en Afrique de l'Est il y a environ 18 600 ans. E-M78 aurait migré hors d'Afrique via l'Égypte à la fin de la période glaciaire il y a environ 11 000 ans pour coloniser le Levant, l'Anatolie et la Grèce.
E-M78 est divisé en 4 branches principales : E-V12, E-V13, E-V22 et E-V65. En Europe, on retrouve surtout le sous-groupe E-V13 principalement en Albanie, Grèce (40 %) et en Sicile.
Bien qu’il s’agisse de l’un des haplogroupes les plus fréquents dans les populations modernes des Balkans, les origines de l’E-V13 sont énigmatiques. La première mention de cet haplogroupe parmi les groupes historiquement attestés se trouve en Bulgarie au cours de de l’âge du bronze-âge du fer, suggérant une association avec le peuple connu sous le nom de « Thraces »[9]. Au début de l'ère romaine, l'E-V13 a probablement connu une augmentation démographique significative, car il apparaît à des fréquences moyennes à élevées dans des régions où, au cours des âges du bronze et du fer précédents, il était soit très rare (Croatie, Hongrie), soit totalement absent (Serbie). Cette association de l'expansion de l'E-V13 avec les populations du sud-est des Balkans du monde thrace semble corroborer les documents historiques de la fin de l’Empire romain qui mentionnent la présence de groupes « thraces » connus sous le nom de « Bessi » dans tous les Balkans jusqu'au VIe siècle de notre ère[9].
E-PF2431
E-PF2431 (E1b1b1b1b), dont l'âge est estimé aux alentours de 13 700 ans[10], a été découvert à Sandorfalva-eperjes en Hongrie, il s'agit d'un conquérant hongrois qui a vécu au Xe siècle[11], ainsi que sur les sites médiévaux de Villa Magna en Italie (XIVe siècle)[12] et de Ellwangen en Allemagne (XVIe siècle)[13]. Il est à noter que nous trouvons E-PF2431, principalement, concentré dans le Derbyshire en Angleterre, dans le Souss au Maroc et dans l'Est de l'Algérie[14].
E–M81
[modifier | modifier le code]E–M81 (E1b1b1b1a), datant d'environ 13 700 ans[15], a été retrouvé dans des vestiges andalous (VIIe – XIIIe siècle) en Espagne et en France[16],[17],[18]. E–M81 se trouve aujourd'hui principalement au Maghreb (où sa fréquence moyenne est > 50 %)[19] et culmine parmi des communautés tunisiennes avec des fréquences atteignant 100 %[20].
E-M183
[modifier | modifier le code]E-M183 (anciennement E1b1b1b), principal sous-clade de E-M81, a une origine locale, nord ouest africaine[21]. E-M183 (E-M81) est la lignée paternelle la plus fréquente en Afrique du Nord. Une étude de 2017 révèle une forte ressemblance entre les haplotypes E-M183 Y-STR ce qui indique une expansion rapide de cet haplogroupe. Les estimations mises à jour de l'ancêtre commun le plus récent (TMRCA) pour E-M183 mettent en évidence une origine extrêmement récente de cet haplogroupe (2 000 à 3 000 ans). Selon cette étude, il n'est pas possible d'écarter une expansion venue du Proche-Orient, qui aurait pu être générée par l'expansion islamique du VIIe siècle, mais certainement pas par celle du Néolithique, apparu en Afrique du Nord-Ouest vers 7400 avant présent et qui pourrait avoir présenté une forte persistance épipaléolithique. Une telle apparition récente de E-M183 dans le nord-ouest de l'Afrique cadrerait avec les tendances observées dans le reste du génome, où un mélange étendu, notamment chez les hommes venant du Proche-Orient, est enregistré il y a près de 1 300 ans, coïncidant avec l'expansion arabe[22].
Une autre hypothèse impliquerait que l'E-M183 a été créé quelque part en Afrique du Nord-Ouest puis s'est ensuite répandu dans toute la région. Les estimations temporelles concernant l'origine de cet haplogroupe se chevauchent avec la fin de la troisième guerre punique (146 av. J.-C.), lorsque Carthage (dans l'actuelle Tunisie) fut vaincue et détruite, marquant la dispersion de la noblesse carthaginoise dans le reste Afrique du Nord. Il y a environ 2 000 ans, l'Afrique du Nord était l'une des provinces les plus riches de Rome et E-M183 pourrait avoir connu la croissance démographique résultante[22].
Il a notamment été retrouvé chez deux individus d'une nécropole islamique de la ville de Valence, datant du XIIe – XIIIe siècle[23].
La fréquence de E-M183 se situe généralement autour de 50 % en Afrique du Nord mais atteint parfois 100 % dans de nombreuses populations non-métissées. En Europe, cet haplogroupe se retrouve surtout dans la péninsule Ibérique, où sa fréquence dépasse parfois les 10 % avec la plus forte concentration dans le sud et le nord de l'Espagne, où sa fréquence atteint 40 % chez les Pasiegos et les Andalous[24],[25]. Il est aussi présent au Moyen-Orient, en Italie et dans le Sud de la France.
E-M34
[modifier | modifier le code]E-M34 (anciennement E1b1b1c) caractérisé par les marqueurs M123 et M34, rare en Europe, est surtout présent au Moyen-Orient (Arabie Saoudite, Jordanie, Yémen, etc.) et parmi les populations sémites d'Éthiopie. Il est aussi assez fréquent chez les juifs (10 %)[26]. Sa fréquence est également très faible dans le Caucase (< 1 %)[27].
Distribution de E1b1b et de ses trois principaux sous-groupes
[modifier | modifier le code]| Région et Population | N | E-M35* | E-M78 | E-M81 | E-M34 | Source |
|---|---|---|---|---|---|---|
| Europe | ||||||
| Portugais du nord | 50 | 2 | 4 | 4 | … | Cruciani et al. 2004[28] |
| Portugais du sud | 49 | … | 4.1 | 12.2 | … | Cruciani et al. 2004 |
| Pasiegos de Cantabrie | 56 | ... | … | 41.1 | … | Cruciani et al. 2004 |
| Asturiens | 90 | … | 10 | 2.2 | 1.1 | Cruciani et al. 2004 |
| Espagnols du sud | 62 | 1.6 | 3.2 | 1.6 | … | Cruciani et al. 2004 |
| Basques Espagnols | 55 | … | … | 3.6 | … | Cruciani et al. 2004 |
| Français du sud-ouest | 85 | … | 4.7 | 3.5 | … | Cruciani et al. 2004 |
| Basques Français | 16 | … | 6.3 | … | … | Cruciani et al. 2004 |
| Corses | 140 | 0.7 | 4.3 | … | 1.4 | Cruciani et al. 2004 |
| Orcadiens | 7 | … | … | … | … | Cruciani et al. 2004 |
| Danois | 35 | … | 2.9 | … | … | Cruciani et al. 2004 |
| Italiens du nord | 67 | … | 9 | 1.5 | 1.5 | Cruciani et al. 2004 |
| Italiens du centre | 89 | … | 11 | 2.2 | … | Cruciani et al. 2004 |
| Italiens du sud | 87 | … | 11.5 | … | 2.3 | Cruciani et al. 2004 |
| Siciliens | 136 | … | 16 | 0.7 | 6.6 | Cruciani et al. 2004 |
| Sardes | 367 | 1.1 | 3.5 | 0.3 | 3.5 | Cruciani et al. 2004 |
| Polonais | 38 | … | 2.6 | … | … | Cruciani et al. 2004 |
| Estoniens | 74 | 1.4 | 4.1 | … | … | Cruciani et al. 2004 |
| Russes | 42 | … | … | … | … | Cruciani et al. 2004 |
| Roumains | 14 | … | 17.7 | … | … | Cruciani et al. 2004 |
| Bulgares | 116 | … | 18.4 | … | … | Cruciani et al. 2004 |
| Albanais | 19 | … | 31.6 | … | … | Cruciani et al. 2004 |
| Afrique du Nord | ||||||
| Arabes marocains | 54 | … | 38.9 | 31.5 | … | Cruciani et al. 2004 |
| Berbères du Moyen Atlas | 69 | … | 10.1 | 71 | … | Cruciani et al. 2004 |
| Berbères de Marrakech | 29 | 3.4 | 6.9 | 72.4 | 3.4 | Cruciani et al. 2004 |
| Berbères Mozabites | 20 | … | … | 80 | … | Cruciani et al. 2004 |
| Égyptiens du nord | 21 | … | 28.6 | 4.8 | 4.8 | Cruciani et al. 2004 |
| Égyptiens du sud | 34 | … | 17.6 | … | … | Cruciani et al. 2004 |
| Afrique de l'Est | ||||||
| Juifs éthiopiens | 22 | 9.1 | 9.1 | … | 13.6 | Cruciani et al. 2004 |
| Éthiopiens Amhara | 34 | 2.9 | 8.8 | … | 23.5 | Cruciani et al. 2004 |
| Éthiopiens Oromo | 25 | 12 | 32 | … | 8 | Cruciani et al. 2004 |
| Éthiopiens Wolayta | 12 | 16.7 | 16.7 | … | 8.3 | Cruciani et al. 2004 |
| Divers Éthiopiens | 12 | 8.3 | 33.3 | … | … | Cruciani et al. 2004 |
| Somaliens | 23 | 17.4 | 52.2 | … | … | Cruciani et al. 2004 |
| Borana (Oromo) du Kenya | 7 | 14.3 | 71.4 | … | … | Cruciani et al. 2004 |
| Bantu du Kenya | 28 | 10.7 | 3.6 | … | … | Cruciani et al. 2004 |
| Nilo-Sahariens du Kenya | 18 | 11.1 | 11.1 | … | … | Cruciani et al. 2004 |
| Afrique sub-saharienne | Cruciani et al. 2004 | |||||
| Sénégalais Mandenka | 16 | … | 6.3 | … | … | Cruciani et al. 2004 |
| Songhai du Niger | 10 | … | … | … | … | Cruciani et al. 2004 |
| Touareg du Niger | 22 | … | 4.5 | 9.1 | … | Cruciani et al. 2004 |
| Fulbe du Niger | 7 | … | … | … | … | Cruciani et al. 2004 |
| Fulbe du Nigeria | 32 | … | … | … | … | Cruciani et al. 2004 |
| Hausa du Nigeria | 10 | … | … | … | … | Cruciani et al. 2004 |
| Yoruba du Nigeria | 21 | … | … | … | … | Cruciani et al. 2004 |
| Pygmées Biaka | 33 | … | … | … | … | Cruciani et al. 2004 |
| Pygmées Mbuti | 13 | … | … | … | … | Cruciani et al. 2004 |
| San de Namibie | 7 | … | … | … | … | Cruciani et al. 2004 |
| Africains du sud Kung | 64 | 10.9 | … | … | … | Cruciani et al. 2004 |
| Africains du sud Khwe | 26 | 30.8 | … | … | … | Cruciani et al. 2004 |
| Africains du sud Bantous | 8 | 12.5 | … | … | … | Cruciani et al. 2004 |
| Moyen-Orient | Cruciani et al. 2004 | |||||
| Sépharades de Turquie | 19 | … | … | 5.3 | 5.3 | Cruciani et al. 2004 |
| Turcs d'Istanbul | 35 | … | 5.6 | 3.7 | 2.9 | Cruciani et al. 2004 |
| Turcs du sud-ouest | 40 | … | 8.5 | 2.5 | 2.5 | Cruciani et al. 2004 |
| Turcs du nord-est | 41 | … | … | 2.4 | … | Cruciani et al. 2004 |
| Anatoliens du centre | 61 | … | 6.6 | … | 3.3 | Cruciani et al. 2004 |
| Turcs du sud-est | 24 | … | 4.2 | … | 4.2 | Cruciani et al. 2004 |
| Turcs d'Erzurum | 25 | … | 4 | … | 8 | Cruciani et al. 2004 |
| Turcs Cypriotes | 46 | … | 13 | 8.7 | 2.2 | Cruciani et al. 2004 |
| Bedouins | 28 | … | 3.6 | 3.6 | 7.1 | Cruciani et al. 2004 |
| Arabes Druzes | 28 | … | 10.7 | … | 3.6 | Cruciani et al. 2004 |
| Palestiniens | 29 | … | 10.3 | … | 3.4 | Cruciani et al. 2004 |
| Arabes des Émirats arabes unis | 41 | … | 2.4 | … | 4.9 | Cruciani et al. 2004 |
| Omanites | 13 | … | 7.7 | … | 7.7 | Cruciani et al. 2004 |
| Caucase | Cruciani et al. 2004 | |||||
| Azéris | 97 | … | 2.1 | … | 2.1 | Cruciani et al. 2004 |
| Adygei | 18 | … | … | … | … | Cruciani et al. 2004 |
| Pakistanais | 176 | … | 1.1 | … | … | Cruciani et al. 2004 |
| Est-Asiatiques | 245 | … | … | … | … | Cruciani et al. 2004 |
| Océaniens | 21 | … | … | … | … | Cruciani et al. 2004 |
| Amérindiens | 43 | … | … | … | … | Cruciani et al. 2004 |
E2
[modifier | modifier le code]E2 (M75) présent à la fois dans l'Est et l'Ouest de l'Afrique.
Personnalités appartenant à l'haplogroupe E
[modifier | modifier le code]E1b1a
[modifier | modifier le code]E1b1b
[modifier | modifier le code]- Aaron: en analysant l'ADN des prêtres samaritains, il se pourrait que Moïse ait appartenu à l'haplogroupe 'E1b1b'[29].
- Napoléon Ier : selon une analyse génétique de Gérard Lucotte l'haplogroupe du chromosome Y (ADN-Y) de Napoléon Bonaparte est E-M34 (anciennement E1b1)[30].
- Adolf Hitler : en analysant l'ADN des membres de la famille d'Hitler, il se pourrait qu'Hitler ait appartenu à l'haplogroupe 'E1b1b', peu présent en Allemagne et en Europe occidentale. Cette « empreinte génétique » est beaucoup plus répandue dans le Sud de l'Europe chez 25 % des Grecs et des Siciliens et chez pas moins de 50 à 80 % des Nord-Africains[31],[32].
- William Harvey
- Orville et Wilbur Wright
- Albert Einstein
Références
[modifier | modifier le code]- Kamin M, Saag L, Vincente M, et al., « A recent bottleneck of Y chromosome diversity coincides with a global change in culture », Genome Research, vol. 25, no 4, , p. 459–466 (PMID 25770088, PMCID 4381518, DOI 10.1101/gr.186684.114)
- (en) Haber M, Jones AL, Connel BA, Asan, Arciero E, Huanming Y, Thomas MG, Xue Y, Tyler-Smith C, « A Rare Deep-Rooting D0 African Y-chromosomal Haplogroup and its Implications for the Expansion of Modern Humans Out of Africa », Genetics, , genetics.302368.2019 (PMID 31196864, DOI 10.1534/genetics.119.302368, lire en ligne)
- « Haplogroupe E1b1b (Y-ADN) », sur Eupedia (consulté le ).
- (en) P. A. Underhill, G. Passarino, A. A. Lin, P. Shen, M. Mirazon Lahr, R. A. Foley, P. J. Oefner et L. L. Cavalli-Sforza, « The phylogeography of Y chromosome binary haplotypes and the origins of modern human populations », Annals of Human Genetics, vol. 65, no Pt 1, , p. 43–62 (PMID 11415522, DOI 10.1046/j.1469-1809.2001.6510043.x)
- T. M. Karafet, F. L. Mendez, M. B. Meilerman, P. A. Underhill, S. L. Zegura et M. F. Hammer, « New binary polymorphisms reshape and increase resolution of the human Y chromosomal haplogroup tree », Genome Research, vol. 18, no 5, , p. 830–8 (PMID 18385274, PMCID 2336805, DOI 10.1101/gr.7172008)
- (en) G David Poznik, « Punctuated bursts in human male demography inferred from 1,244 worldwide Y-chromosome sequences », Nature Genetics, vol. 48, no 6, , p. 593–599 (PMID 27111036, PMCID 4884158, DOI 10.1038/ng.3559)
- Trombetta et al. 2015, Phylogeographic refinement and large scale genotyping of human Y chromosome haplogroup E provide new insights into the dispersal of early pastoralists in the African continent
- (en) Vicente M Cabrera, Patricia Marrero, Khaled K Abu-Amero et Jose M Larruga, « Carriers of mitochondrial DNA macrohaplogroup L3 basic lineages migrated back to Africa from Asia around 70,000 years ago », BMC evolutionary biology, vol. 18, no 1 (98), (DOI 10.1186/s12862-018-1211-4, lire en ligne)
- (en) Leonidas-Romanos Davranoglou, Aris Aristodemou, David Wesolowski et Alexandros Heraclides, Ancient DNA reveals the origins of the Albanians, biorxiv.org, 7 juin 2023, doi.org/10.1101/2023.06.05.543790
- « E-PF2431 YTree », sur www.yfull.com (consulté le )
- (en) Zoltán Maróti, Endre Neparáczki, Oszkár Schütz et Kitti Maár, « The genetic origin of Huns, Avars, and conquering Hungarians », Current Biology, vol. 32, no 13, , p. 2858–2870.e7 (ISSN 0960-9822, DOI 10.1016/j.cub.2022.04.093, lire en ligne, consulté le )
- (en) Margaret L. Antonio, Ziyue Gao, Hannah M. Moots et Michaela Lucci, « Ancient Rome: A genetic crossroads of Europe and the Mediterranean », Science, vol. 366, no 6466, , p. 708–714 (ISSN 0036-8075 et 1095-9203, PMID 31699931, PMCID PMC7093155, DOI 10.1126/science.aay6826, lire en ligne, consulté le )
- Alexander Immel, Felix M. Key, András Szolek et Rodrigo Barquera, « Analysis of Genomic DNA from Medieval Plague Victims Suggests Long-Term Effect of Yersinia pestis on Human Immunity Genes », Molecular Biology and Evolution, vol. 38, no 10, , p. 4059–4076 (ISSN 1537-1719, PMID 34002224, PMCID 8476174, DOI 10.1093/molbev/msab147, lire en ligne, consulté le )
- (en) « FamilyTreeDNA - Genetic Testing for Ancestry, Family History & Genealogy », sur www.familytreedna.com (consulté le )
- « E-M81 YTree », sur www.yfull.com (consulté le )
- (en) Marina Silva, Gonzalo Oteo-García, Rui Martiniano et João Guimarães, « Biomolecular insights into North African-related ancestry, mobility and diet in eleventh-century Al-Andalus », Scientific Reports, vol. 11, no 1, , p. 18121 (ISSN 2045-2322, DOI 10.1038/s41598-021-95996-3, lire en ligne, consulté le )
- Iñigo Olalde, Swapan Mallick, Nick Patterson et Nadin Rohland, « The genomic history of the Iberian Peninsula over the past 8000 years », Science (New York, N.Y.), vol. 363, no 6432, , p. 1230–1234 (ISSN 0036-8075, PMID 30872528, PMCID 6436108, DOI 10.1126/science.aav4040, lire en ligne, consulté le )
- Yves Gleize, Fanny Mendisco, Marie-Hélène Pemonge et Christophe Hubert, « Early Medieval Muslim Graves in France: First Archaeological, Anthropological and Palaeogenomic Evidence », PLoS ONE, (DOI 10.1371/journal.pone.0148583, lire en ligne, consulté le )
- (en) Asmahan Bekada, Rosa Fregel, Vicente M. Cabrera et José M. Larruga, « Introducing the Algerian Mitochondrial DNA and Y-Chromosome Profiles into the North African Landscape », PLOS ONE, vol. 8, no 2, , e56775 (ISSN 1932-6203, PMID 23431392, PMCID PMC3576335, DOI 10.1371/journal.pone.0056775, lire en ligne, consulté le )
- (en) Sarra Elkamel, Sofia L. Marques, Luis Alvarez et Veronica Gomes, « Insights into the Middle Eastern paternal genetic pool in Tunisia: high prevalence of T-M70 haplogroup in an Arab population », Scientific Reports, vol. 11, no 1, , p. 15728 (ISSN 2045-2322, DOI 10.1038/s41598-021-95144-x, lire en ligne, consulté le )
- Neus Solé-Morata, Carla García-Fernández, Vadim Urasin et Asmahan Bekada, « Whole Y-chromosome sequences reveal an extremely recent origin of the most common North African paternal lineage E-M183 (M81) », Scientific Reports, vol. 7, , p. 15941 (ISSN 2045-2322, PMID 29162904, PMCID 5698413, DOI 10.1038/s41598-017-16271-y, lire en ligne, consulté le )
- (en) Neus Solé-Morata, Carla García-Fernández, Vadim Urasin..., Whole Y-chromosome sequences reveal an extremely recent origin of the most common North African paternal lineage E-M183 (M81), Scientific Reports, volume 7, Article number: 15941. 2017
- (en) Marina Silva, Gonzalo Oteo-García, Rui Martiniano et al., Biomolecular insights into North African-related ancestry, mobility and diet in eleventh-century Al-Andalus, Scientific Reports, volume 11, Article numéro: 18121, 2021, doi.org/10.1038/s41598-021-95996-3
- R. Scozzari, F. Cruciani, A. Pangrazio et P. Santolamazza, « Human Y-chromosome variation in the western Mediterranean area: implications for the peopling of the region », Human Immunology, vol. 62, no 9, , p. 871–884 (ISSN 0198-8859, PMID 11543889, DOI 10.1016/s0198-8859(01)00286-5, lire en ligne, consulté le )
- G. Lucotte, N. Gérard et G. Mercier, « North African genes in Iberia studied by Y-chromosome DNA haplotype V », Human Immunology, vol. 62, no 9, , p. 885–888 (ISSN 0198-8859, PMID 11543890, DOI 10.1016/s0198-8859(01)00287-7, lire en ligne, consulté le )
- (en) Selina Brace, Yoan Diekmann, Thomas Booth et al., Genomes from a medieval mass burial show Ashkenazi-associated hereditary diseases pre-date the 12th century, Current Biology, 30 août 2022, doi.org/10.1016/j.cub.2022.08.036
- The Caucasus as an asymmetric semipermeable barrier to ancient human migrations, Bayazit Yunusbayev et al. 2011
- Phylogeographic analysis of haplogroup E3b (E-M215) y chromosomes reveals multiple migratory events within and out of Africa, Cruciani F, La Fratta R, Santolamazza P, Sellitto D, Pascone R, Moral P, Watson E, Guida V, Colomb EB, Zaharova B, Lavinha J, Vona G, Aman R, Cali F, Akar N, Richards M, Torroni A, Novelletto A, Scozzari R Source Dipartimento di Genetica e Biologia Molecolare, Universita "La Sapienza", 00185 Rome, Italy Am J Hum Genet. 2004 May;74(5):1014-22. Epub 2004 Mar 24.
- Peter J. Oefner, Georg Hölzi, Piedong Shen et Isaac Shpirer, « Genetics and the history of the Samaritans: Y-chromosomal microsatellites and genetic affinity between Samaritans and Cohanim », Human Biology, vol. 85, no 6, , p. 825–858 (ISSN 1534-6617, PMID 25079122, DOI 10.3378/027.085.0601, lire en ligne, consulté le )
- Haplogroup of the Y Chromosome of Napoléon the First, Gerard Lucotte, Thierry Thomasset, Peter Hrechdakian, janvier 2012]
- Hitler was verwant met Somaliërs, Berbers en Joden, Knack, 18 août 2010
- Hitler verwant met Somaliërs, Berbers en Joden, De Standaard, 18 août 2010
Bibliographie
[modifier | modifier le code]- B. Arredi et al.: A Predominantly Neolithic Origin for Y-Chromosomal DNA Variation in North Africa. American Journal Of Human Genetics, 2004, p. 338–345
- F. Cruciani et al.: A Back Migration from Asia to Sub-Saharan Africa Is Supported by High-Resolution Analysis of Human Y-Chromosome Haplotypes. American Journal Of Human Genetics, 2002, p. 1197–1214
- F. Cruciani et al.: Phylogeographic Analysis of Haplogroup E3b (E-M215) Y Chromosomes Reveals Multiple Migratory Events Within and Out Of Africa. American Journal Of Human Genetics, 2004, p. 1014–1022
- F. Cruciani et al.: Molecular Dissection of the Y Chromosome Haplogroup M-78
- J. R. Luis et al.: The Levant versus the Horn of Africa: Evidence for Bidirectional Corridors of Human Migrations. American Journal Of Human Genetics, 2004, p. 523-544
- J. J. Sanchez et al.: High frequencies of Y chromosome lineages characterized by E3b1, DYS19-11, DYS392-12 in Somali males. European Journal of Human Genetics, 2005, p. 856–86
- A. Salas et al.: The Making of the African mtDNA Landscape. American Journal Of Human Genetics, 2002, p. 1082–1111
- O. Semino et al.: Origin, Diffusion, and Differentiation of Y-Chromosome Haplogroups E and J: Inferences on the Neolithization of Europe and Later Migratory Events in the Mediterranean Area, American Journal of Human Genetics, 2004, p. 1023-1034
- E. T. Wood et al.: Contrasting patterns of Y chromosome and mtDNA variation in Africa: evidence for sex-biased demographic processes, European Journal of Human Genetics, 2005, p. 867–876
Liens externes
[modifier | modifier le code]|
Haplogroupes du chromosome Y (Y-ADN) | ||||||||||||||||||||||||
| Plus récent ancêtre patrilinéaire commun | ||||||||||||||||||||||||
| A | ||||||||||||||||||||||||
| BT | ||||||||||||||||||||||||
| B | CT | |||||||||||||||||||||||
| DE | CF | |||||||||||||||||||||||
| D | E | C | F | |||||||||||||||||||||
| G | H | IJK | ||||||||||||||||||||||
| IJ | K | |||||||||||||||||||||||
| I | J | LT | K2 | |||||||||||||||||||||
| I1 | L | T | MS | P | NO | |||||||||||||||||||
| M | S | Q | R | N | O | |||||||||||||||||||
| R1 | R2 | |||||||||||||||||||||||
| R1a | R1b | |||||||||||||||||||||||