Bacteriófago


Los bacteriófagos (también llamados fagos, del griego φαγητόν faguētón, ‘alimento’, ‘ingestión’) son virus que infectan exclusivamente a las bacterias.
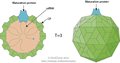
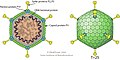
Al igual que los virus que infectan células eucariotas, los fagos están constituidos por una cubierta proteica o cápside en cuyo interior está contenido su material genético, que puede ser ADN, de 5000 a 500 000 pares de bases. El tamaño de los fagos oscila entre 20 y 200 nm aproximadamente.[1]
Se pueden dividir en bacteriófagos de "cola" y sin "cola". La diferencia entre ambos es que los bacteriófagos de cola poseen unas pinzas que permiten inyectar el material genético dentro la bacteria huésped y no dependen del ingreso dentro la célula huésped. En contraste los bacteriófagos sin cola son similares a los virus eucariotas porque dependen ingresar dentro la célula huésped para su replicación. Entre los bacteriófagos sin cola se pueden hallar los bacteriófagos filamentosos clasificados en el reino Loebvirae y bacteriófagos icosaedros o esféricos. Los bacteriófagos de cola comprenden más del 80 % de los fagos y se clasifican en la clase Caudoviricetes.[2] Los bacteriófagos sin cola icosaedros o esféricos se clasifican en diversos taxones virales.
Pueden ser de ADN o ARN, sin embargo no se han detectado bacteriófagos de retrotranscripción, es decir que se repliquen por una cadena intermediaria de material genético diferente del que están compuestos como los retrovirus. Los bacteriófagos de cola son exclusivamente de ADN.
Los fagos son ubicuos y pueden ser encontrados en diversos tipos de bacterias, tanto en el suelo como en la población de microorganismos del intestino de los animales. Uno de los ambientes más poblados por fagos y otros virus es el agua de mar, donde se estima que puede haber en torno a 109 partículas virales por mililitro, pudiendo estar infectadas por fagos el 70 % de las bacterias marinas.[3]
Clasificación
[editar]Los bacteriófagos se pueden hallar en todos los dominios o linajes virales establecidos y constituyen las formas ancestrales de los virus eucariotas de dichos linajes. Los bacteriófagos descritos hasta el momento tienen genomas (de ADN bicatenario, ADN monocatenario, ARN bicatenario y ARN monocatenario positivo o negativo,) según la clasificación de Baltimore. Sin embargo, no se detectaron bacteriófagos con genomas de (ARN monocatenario retrotranscrito y ADN bicatenario retrotranscrito). Esto no quiere decir que no existan bacteriófagos con genomas de (ARN monocatenario negativo, ARN monocatenario retrotranscrito y ADN bicatenario retrotranscrito) según la clasificación de Baltimore; alternativamente, pueden existir bacteriófagos con estos genomas y que todavía no hayan sido descubiertos. Los bacteriófagos pueden tener viriones con diferentes formas, aunque los más abundantes y populares son los bacteriófagos de cola.[4] Los bacteriófagos y los arqueófagos que infectan arqueas se pueden distinguir de los virus eucariotas por la presencia de las secuencias de Shine-Dalgarno típicas de procariotas en sus genomas, lo cual es una herramienta que permite la identificación de nuevos bacteriófagos o virus procariotas.[5]
Según investigaciones filogenéticas la mayoría de los bacteriófagos se originaron antes que el último antepasado común universal y por tanto infectaron al último antepasado común universal y a los protobiontes, (estructuras lipidícas compuestas por material genético que precedieron a las células). El origen de los bacteriófagos junto con los arqueofagos que son los primeros virus es incierto, se ha sugerido que se originaron en los protobiontes durante el mundo de ARN.[6] En el momento del origen de los eucariotas los bacteriófagos o arqueofagos precederían a varios virus que actualmente infectan a los eucariotas. Por ejemplo los bacteriófagos de cola (Caudoviricetes) son ancestros de los Herpesvirales que incluye a los virus del herpes y la varicela.
Los estudios de metagenómica han revelado la existencia de 189,680 especies de bacteriófagos de ADN nuevos principalmente de cola en la microbiota humana, así como la presencia de millones de especies en diversos ecosistemas.[7][8] Y la existencia de múltiples taxones nuevos de bacteriófagos de ARN previamente desconocidos en diversos ecosistemas, incluyendo millones de especies.[9]
Los bacteriófagos actualmente se clasifican en los siguientes taxones según ICTV (2022):
- Dominio Duplodnaviria
- Clase Caudoviricetes (bacteriófagos de cola, ADN bicatenario)[4]
- Dominio Monodnaviria
- Reino Sangervirae (bacteriófagos icosaedros, ADN monocatenario)[4]
- Reino Loebvirae (bacteriófagos filamentosos, ADN monocatenario)[4]
- Dominio Varidnaviria
- Reino Helvetiavirae (bacteriófagos icosaedros, ADN bicatenario)[4]
- Reino Bamfordvirae
- Filo Preplasmiviricota
- Clase Tectiliviricetes (bacteriófagos icosaedros, ADN bicatenario)[4]
- Familia Finnlakeviridae (bacteriófagos icosaedros, ADN monocatenario)[4]
- Filo Preplasmiviricota
- Bacteriófagos de colocación incierta:
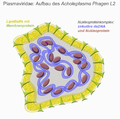
- Familia Plasmaviridae (bacteriófagos pleomorficos, ADN bicatenario)[4]
- Dominio Riboviria
- Filo Duplornaviricota
- Familia Cystoviridae (bacteriófagos icosaedros, ARN bicatenario)[4]
- Filo Lenarviricota
- Clase Leviviricetes (bacteriófagos icosaedros, ARN monocatenario positivo)[4]
- Filo Pisuviricota
- Familia Picobirnaviridae (bacteriófagos icosaedros, ARN bicatenario)[9]
- Familia Partitiviridae (bacteriófagos icosaedros, ARN bicatenario)[9]
- Filo Artimaviricota (bacteriófagos icosaedros, ARN bicatenario)[10]
- Filo Taraviricota (bacteriófagos, ARN monocatenario positivo o ARN bicatenario)[11]
- Filo Duplornaviricota
Galería
[editar]Historia
[editar]La historia del descubrimiento de los bacteriófagos ha sido objeto de muchos debates, los cuales incluyen una controversia sobre quién fue su descubridor. En 1913, el bacteriólogo británico Frederick Twort descubrió un agente bacteriolítico que infectaba y mataba a las bacterias, pero, no fue capaz de saber qué era exactamente dicho agente, señalando en una de sus hipótesis que, entre otras posibilidades, podría tratarse de un virus. No fue sino hasta 1917, cuando el microbiólogo canadiense Félix d'Herelle anunció el descubrimiento de "un invisible antagonista microbiano del bacilo de la disentería", al cual afirmó que se trataba de un virus al cual llamó bacteriófago. Los trabajos de d'Herelle iniciaron desde 1910 y en contraste con Twort y algunos otros científicos que habían reportado fenómenos similares, tuvo pocas dudas sobre la naturaleza del fenómeno que estaba observando y afirmó que se trataba de un virus que parasitaba a la bacteria. El nombre bacteriófago lo formó de la palabra "bacteria" y "phagein" (comer o devorar, en griego), implicando que los fagos "comen" o "devoran" a las bacterias.
Biología
[editar]Los fagos se pueden dividir en virulentos y temperados en función de su ciclo de vida. Los fagos virulentos producen el ciclo lítico, en este ciclo se unen a su huésped bacteriano, inyectan su genoma, se reproducen gracias a la maquinaria molecular del huésped y, finalmente, lisan a la célula huésped, liberando al mismo tiempo su progenie.[12] El segundo tipo de fagos, los temperados, tienen un estilo de vida diferente e infectan a su huésped mediante el inicio de un ciclo lisogénico, donde el genoma del fago permanece latente como un profago, se replica junto con su huésped y de vez en cuando estalla en un ciclo lítico bajo un disparador específico.[12]
Pueden ser beneficiosos para las bacterias, ya que pueden codificar genes para la resistencia a los antibióticos o de otros factores de virulencia.[13]
Replicación
[editar]Los fagos pueden generar el ciclo lítico o el ciclo lisogénico, aunque muy pocos son capaces de llevar a cabo ambos. Si se lleva a cabo la lisis, no puede llevarse a cabo la lisogenia y viceversa. En el ciclo lítico, las células hospedadoras del fago son lisadas (destruidas) tras la replicación y encapsulación de las partículas virales, de forma que los nuevos virus quedan libres para llevar a cabo una nueva infección.
Por el contrario, en el ciclo lisogénico no se produce la lisis inmediata de la célula. El genoma del fago puede integrarse en el ADN cromosómico de la bacteria hospedadora, replicándose a la vez que lo hace la bacteria o bien puede mantenerse estable en forma de plásmido, replicándose de forma independiente a la replicación bacteriana. En cualquier caso, el genoma del fago se transmitirá a toda la progenie de la bacteria originalmente infectada. El fago queda así en estado de latencia hasta que las condiciones del medio se vean deterioradas: disminución de nutrientes, aumento de agentes mutagénicos, etc. En este instante, los fagos endógenos o profagos se activan y dan lugar al ciclo lítico que termina con la lisis celular.
Conversión lisogénica
[editar]En ocasiones, los profagos otorgan beneficios a la bacteria huésped mientras permanecen en estado letárgico al incorporarle nuevas funciones a su genoma; éste fenómeno se conoce las condiciones de vida de la bacteria.
Acoplamiento
[editar]Los fagos se acoplan a receptores específicos en la superficie de la bacteria, que pueden ser lipopolisacáridos, ácidos teicoicos, proteínas o incluso flagelos. Por ello, cada fago solo podrá infectar ciertas bacterias según sus receptores. Puesto que los fagos no son móviles, dependen de encuentros al azar con los receptores adecuados en solución para poder infectar una bacteria. Tras el reconocimiento del receptor adecuado, el fago queda acoplado a la superficie celular. El material genético puede ser ahora introducido a través de la membrana o bien simplemente depositado sobre la superficie.
Síntesis de proteínas y ácidos nucleicos
[editar]En un corto período, que puede llegar a ser de minutos, los ribosomas bacterianos comienzan a traducir el ARNm viral a proteínas. En el caso de los fagos basados en ARN, una ARN replicasa es sintetizada al inicio del proceso.
- Las proteínas producidas en la fase temprana y unas pocas proteínas que estaban presentes en el virión podrían modificar la ARN polimerasa bacteriana de forma que transcriba preferentemente los ARNm virales. Todo el sistema de traducción y de replicación normal de la bacteria se ve interrumpido y es forzado a producir nuevas partículas virales.
- Posteriormente, las proteínas helper se encargarán de ensamblar las nuevas partículas virales.
- Finalmente, se sintetizan las proteínas de la fase tardía, involucradas en el proceso de la lisis celular.
Ensamblaje
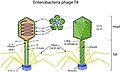
[editar]En el caso del fago T4 con cola, la construcción de nuevas partículas virales es un complejo proceso que requiere la ayuda de ciertas moléculas. La cola y la cabeza o cápside del fago son construidas por separado y se ensamblan posteriormente de forma espontánea solamente en la lisis completa. Después, el ADN es empaquetado en el interior de la cápside mediante un mecanismo no muy bien conocido aún. Todo el proceso puede durar unos 15 minutos. La cabeza tiene simetría icosaédrica (un icosaedro con un prisma hexagonal intercalado). La cola es de simetría helicoidal formada por un tubo central rígido y una vaina contráctil. De la placa basal salen seis espículas basales y seis fibras caudales. En la conexión de la cabeza y la cola existe un collar. Cada una de estas estructuras está formada por diferentes proteínas.
Liberación de los fagos
[editar]Los fagos pueden ser liberados mediante lisis celular o por secreción celular. En el caso del fago T4, unos 20 minutos después de inyectar el material genético, más de 300 fagos son liberados vía lisis. La proteína que lleva a cabo la lisis es la endolisina, una enzima capaz de romper las moléculas de peptidoglicano de la pared bacteriana. Sin embargo, algunos fagos pueden quedarse en la célula como parásitos, de forma que la bacteria va secretando constantemente nuevas partículas virales. En estos casos, los viriones salen mediante procesos de exocitosis, en los que cada uno se queda con una pequeña porción de membrana bacteriana que los envuelve. Todos los nuevos fagos liberados quedan en disposición de infectar a una nueva bacteria.
Biología molecular
[editar]Los fagos cumplen un papel de gran importancia en la biología molecular al ser utilizados como vectores de clonación para insertar ADN dentro de las bacterias y obtener como resultado bibliotecas genómicas. Uno de los fagos más utilizados con este fin es el Fago λ.
Terapia fágica
[editar]Tras el descubrimiento de bacteriófagos, la idea de la terapia de fagos fue tomada con entusiasmo y se puso en práctica, pero su aplicación se calmó posteriormente después del descubrimiento de los antibióticos y su uso en la Segunda Guerra Mundial, floreciente sólo por detrás de la “cortina de hierro”.[14]
La terapia fágica ha sido utilizada desde la década de 1940 en la ex Unión Soviética como una alternativa a los antibióticos para tratar infecciones bacterianas, ya que eliminar bacterias es lo que los fagos hacen mejor.
La mayor parte del conocimiento histórico acerca de la práctica de la terapia de fagos se asocia con dos lugares: el Instituto Hirszfeld en Wroclaw, Polonia y el Instituto Eliava en Tiflis, Georgia. El foco principal del Instituto Eliava ha sido la producción y el uso terapéutico de los cócteles de fagos (por ejemplo pyophage e intestiphage), que se dirigen a bacterias patógenas específicas. Por otra parte, el Instituto Hirszfeld apoya el desarrollo de un enfoque más individualizado en fagos preparaciones terapéuticas. Ambos Institutos tienen una historia de la publicación de las tasas de éxito convincentes de la terapia de fagos, pero sin la corroboración de ensayos clínicos adecuadamente controlados.[15] El desarrollo de cepas bacterianas resistentes a múltiples drogas ha conducido a investigadores en medicina a reconsiderar a los fagos como una alternativa al uso de antibióticos. Hay una biblioteca de búsqueda de fagos específicos y sus usos terapéuticos en el Instituto Tiflis, en la República de Georgia.
En agosto de 2006, la FDA (Food and Drug Administration) de Estados Unidos aprobó el uso de bacteriófagos en ciertas carnes con el fin de acabar con la bacteria Listeria monocytogenes. En el otoño de 2011 la Fundación Bill y Melinda Gates anuncia que financiará la investigación, llevada a cabo por el Centro Nacional de Biotecnología (España), dependiente del CSIC, del uso de bacteriófagos como sustituto de los antibióticos ante las infecciones del tracto digestivo producidas por bacterias.
Elección de bacteriófagos y preparación para fines terapéuticos
[editar]Los bacteriófagos se pueden aislar para fines terapéuticos a partir de cualquier fuente ambiental, en el que es probable que se contenía el patógeno diana. Las aguas residuales probablemente son la fuente más rica de fagos. La forma más directa para aislar un bacteriófago es esterilizar una muestra ambiental con el fin de eliminar los microorganismos no deseados y luego inocularlo en una placa de cultivo. Después del aislamiento, un bacteriófago debe caracterizarse y secuenciarse con el fin de ser utilizado con fines terapéuticos.[16]
Los criterios más importantes para la selección del bacteriófago adecuado son la especificidad del fago, la eficacia y la evitación de efectos adversos.[17] Por esa razón, el bacteriófago seleccionado debe absorber de manera eficiente y ser lítico para el huésped bacteriano objetivo. El uso de fagos temperados no es apropiado para las preparaciones de fagos terapéuticos, ya que lisogenia aumenta la posibilidad de amplificar la virulencia de bacterias diana o inducir resistencia a los fagos.
El desarrollo de la resistencia a bacteriófagos es una preocupación importante más de terapia de fagos. Además, diferentes cepas de bacterias pueden diferir en su susceptibilidad a fagos. Por lo tanto, la modificación de la gama de huéspedes de una preparación terapéutica de fagos podría tener un gran impacto en su éxito. Para asegurar la gama de huéspedes deseable de una formulación de fago, se han desarrollado las mezclas que contienen dos o más bacteriófagos, los llamados “cócteles de fago”.
Bacteriófagos modelo
[editar]A continuación se presenta una lista de los bacteriófagos más ampliamente estudiados:
- Fago λ - Lisógeno
- Fago T4 (169 a 170 kpb, 200 nm de tamaño)
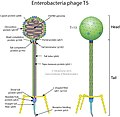
- Fago T7
- Fago M13 - Fagémido
- Fago MS2 (23-25 nm de tamaño)
- Fago Qβ
- Fago φCb5
- Fago Φ6
- Fago P22
- Fago Φ29
- Fago ΦX174
- Fago G4
- Fago P1
- Fago P2
- Fago N4
- Fago R17
Véase también
[editar]Referencias
[editar]- ↑ Stephen., Mc Grath,; van., Sinderen, Douwe (2007). Bacteriophage : genetics and molecular biology. Caister Academic Press. ISBN 9781904455141. OCLC 86168751.
- ↑ Sharp, Richard (2001). «Bacteriophages: biology and history». Journal of Chemical Technology & Biotechnology 76 (7): 667-672. ISSN 0268-2575. doi:10.1002/jctb.434. Consultado el 2 de diciembre de 2018.
- ↑ M., Prescott, Lansing; 1935-, Klein, Donald A., (1993). Microbiology (2nd ed edición). Wm. C. Brown. ISBN 0697013723. OCLC 27302868.
- ↑ a b c d e f g h i j A Roadmap for Genome-Based Phage Taxonomy - MDPI.
- ↑ Krishnamurthy, Siddharth R.; Wang, David (1 de marzo de 2018). «Extensive conservation of prokaryotic ribosomal binding sites in known and novel picobirnaviruses». Virology 516: 108-114. ISSN 0042-6822. PMID 29346073. doi:10.1016/j.virol.2018.01.006.
- ↑ Mart Krupovic, Valerian V. Dolja, Eugene V. Koonin (2020). The LUCA and its complex virome. Nature.
- ↑ Stephen Nayfach, David Páez-Espino, Lee Call, Soo Jen Low, Hila Sberro, Natalia N. Ivanova, Amy D. Proal, Michael A. Fischbach, Ami S. Bhatt, Philip Hugenholtz & Nikos C. Kyrpides (2021). Metagenomic compendium of 189,680 DNA viruses from the human gut microbiome. Nature.
- ↑ Simon Roux, Mart Krupovic, Rebecca A. Daly, Adair L. Borges, Stephen Nayfach, Frederik Schulz, Emiley A. Eloe-Fadrosh et al.: Cryptic inoviruses revealed as pervasive in bacteria and archaea across Earth’s biomes. In: Nature Microbiology, 22 July 2019, doi:10.1038/s41564-019-0510-x
- ↑ a b c Uri Neri, Yuri I. Wolf, Simon Roux, Antonio Pedro Camargo, Darius Kazlauskas, I. Min Chen, Benjamin Lee, Natalia Ivanova, Lisa Zeigler Allen, David Paez-Espino, Donald A. Bryant, Devaki Bhaya, Mart Krupovic, Valerian V. Dolja, Nikos C. Kyrpides, Eugene V. Koonin, Uri Gophna (2022). A five-fold expansion of the global RNA virome reveals multiple new clades of RNA bacteriophages. Biorxiv.
- ↑ Double-stranded RNA sequencing reveals distinct riboviruses associated with thermoacidophilic bacteria from hot springs in Japan.
- ↑ Cryptic and abundant marine viruses at the evolutionary origins of Earth’s RNA virome. Science org.
- ↑ a b Kutter, Elizabeth, ed. (28 de diciembre de 2004). Bacteriophages. doi:10.1201/9780203491751. Consultado el 2 de diciembre de 2018.
- ↑ Lin, Derek M; Koskella, Britt; Lin, Henry C (2017). «Phage therapy: An alternative to antibiotics in the age of multi-drug resistance». World Journal of Gastrointestinal Pharmacology and Therapeutics 8 (3): 162. ISSN 2150-5349. doi:10.4292/wjgpt.v8.i3.162. Consultado el 2 de diciembre de 2018.
- ↑ Abedon, Stephen T.; Kuhl, Sarah J.; Blasdel, Bob G.; Kutter, Elizabeth Martin (2011-03). «Phage treatment of human infections». Bacteriophage 1 (2): 66-85. ISSN 2159-7081. doi:10.4161/bact.1.2.15845. Consultado el 4 de diciembre de 2018.
- ↑ Kutter, Elizabeth; De Vos, Daniel; Gvasalia, Guram; Alavidze, Zemphira; Gogokhia, Lasha; Kuhl, Sarah; Abedon, Stephen (1 de enero de 2010). «Phage Therapy in Clinical Practice: Treatment of Human Infections». Current Pharmaceutical Biotechnology 11 (1): 69-86. ISSN 1389-2010. doi:10.2174/138920110790725401. Consultado el 4 de diciembre de 2018.
- ↑ Joana,, Azeredo,; 1975-, Sillankorva, Sanna,. Bacteriophage therapy : from lab to clinical practice. ISBN 9781493973958. OCLC 1012104696. Consultado el 4 de diciembre de 2018.
- ↑ Weber-Dąbrowska, Beata; Jończyk-Matysiak, Ewa; Żaczek, Maciej; Łobocka, Małgorzata B.; Łusiak-Szelachowska, Marzanna; Górski, Andrzej (9 de noviembre de 2016). «Corrigendum: Bacteriophage Procurement for Therapeutic Purposes». Frontiers in Microbiology 7. ISSN 1664-302X. doi:10.3389/fmicb.2016.01813. Consultado el 4 de diciembre de 2018.
Enlaces externos
[editar] Wikimedia Commons alberga una categoría multimedia sobre Bacteriófago.
Wikimedia Commons alberga una categoría multimedia sobre Bacteriófago.- Phage.org general information on bacteriophages
- Bacteriophages: What are they. Presentation by Professor Graham Hatfull, University of Pittsburgh en YouTube. (en inglés)